Plantilla para análisis del photogate#
En este notebook, dejamos una plantilla para analizar datos tomados con el fotosensor o photogate, y digitalizados por el MotionDAQ.
Si se va a medir para muchas condiciones experimentales,
es recomendable nombrar a los archivos con el nombre de la condición experimental.
Por ejemplo, 101.5cm.txt, si se midió a una longitud de 101.5 cm.
En la sección Paso a paso, mostramos que hace cada linea de código con un ejemplo.
Acá, directamente importamos los paquetes y definimos funciones que encapsulan las distintas etapas del análisis de datos. De todas maneras, muchas lineas están comendatas.
from pathlib import Path
import matplotlib.pyplot as plt
import numpy as np
import pandas as pd
def extrar_longitud(nombre: Path):
"""Extrae el valor de la longitud del nombre del archivo."""
longitud = nombre.stem # nombre del archivo sin extension
longitud = longitud.removesuffix("cm") # borra "cm" del final del nombre
longitud = float(longitud) # lo convierte a un número
return longitud
def cargar_archivo_de_motionDAQ(nombre: Path):
"""Carga el archivo y devuelve un array de numpy,
donde la primer columna corresponde al tiempo,
y las demás al voltaje en cada canal.
Para obtener un array con todas las columnas:
>>> datos = cargar_archivo_de_motionDAQ("archivo1.txt")
Si se sabe la cantidad de columnas,
se pueden obtener por separado como:
>>> col1, col2 = cargar_archivo_de_motionDAQ("archivo1.txt")
"""
# Vamos a usar pandas para leer el archivo.
# pandas añade ciertas cosas utiles sobre numpy,
# pero no nos interesa ahora.
data = pd.read_csv(
nombre,
skiprows=4, # ignora las 4 primeras filas del archivo
delimiter="\t", # las columnas estan separadas por "tab"
decimal=",", # separador decimal de los números (en el archivo de texto)
header=None, # las columnas no tienen "titulo"
)
# extraemos el array de numpy subyacente (.values)
# y lo transponemos (.T)
return data.values.T
def encontrar_tiempos_de_saltos(tiempo, voltaje, *, umbral=2):
"""Encuentra los tiempos donde se produce un cambio de voltaje mayor al umbral."""
diferencia_de_voltaje = np.diff(voltaje)
saltos = np.abs(diferencia_de_voltaje) > umbral
indices = np.nonzero(saltos)
return tiempo[indices]
def calcular_periodos(tiempos, *, paso=4):
"""Calcula los periodos como diferencias de tiempos.
Nota: el paso depende del experimento.
En el caso de periodos del péndulo,
el salto en voltaje correspondiente es uno cada 4.
"""
periodos = tiempos[paso:] - tiempos[:-paso]
return periodos
Buscando los archivos#
Buscamos todos los archivos que terminan con .txt en la carpeta actual.
carpeta = Path() # carpeta actual
archivos = list(carpeta.glob("*.txt")) # lista de archivos que terminan con .txt
len(archivos) # cuantos archivos encontró
2
Analizando un archivo#
Graficar datos crudos#
Cargamos el primer archivo, y graficamos la señal:
archivo = archivos[0] # el primer archivo
t, v = cargar_archivo_de_motionDAQ(archivo)
plt.figure(figsize=(6, 2))
plt.xlabel("Tiempo [s]")
plt.ylabel("Voltaje [V]")
plt.plot(t, v)
[<matplotlib.lines.Line2D at 0x7fd04fcf3d90>]
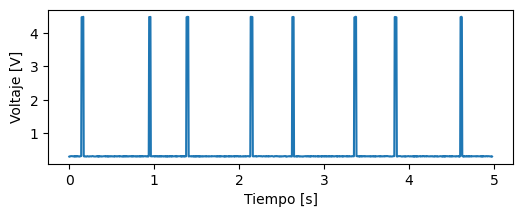
Es importante revisar los archivos, para asegurarse que no haya habido problemas al medir.
Cambio de voltaje#
Vemos que tiempos encuentra en la señal:
archivo = archivos[0] # el primer archivo
t, v = cargar_archivo_de_motionDAQ(archivo)
tiempos = encontrar_tiempos_de_saltos(t, v)
plt.figure(figsize=(6, 2))
plt.xlabel("Tiempo [s]")
plt.ylabel("Voltaje [V]")
plt.plot(t, v, ".--")
plt.vlines(tiempos, 0, 5, color="C1") # lineas verticales
<matplotlib.collections.LineCollection at 0x7fd04f6b1750>
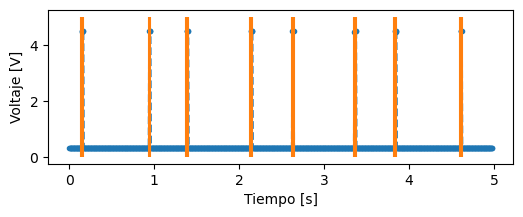
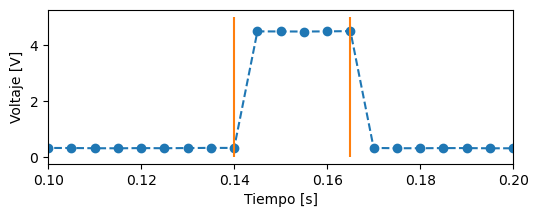
Si hacemos zoom a uno de los saltos:
plt.figure(figsize=(6, 2))
plt.xlabel("Tiempo [s]")
plt.ylabel("Voltaje [V]")
plt.plot(t, v, "o--")
plt.vlines(tiempos, 0, 5, color="C1") # lineas verticales
plt.xlim(0.1, 0.2) # zoom en el eje x
(0.1, 0.2)
Periodos#
Veamos que periodos calcula para la señal:
archivo = archivos[0] # el primer archivo
t, v = cargar_archivo_de_motionDAQ(archivo)
tiempos = encontrar_tiempos_de_saltos(t, v)
periodos = calcular_periodos(tiempos)
plt.figure(figsize=(6, 2))
plt.xlabel("Número de periodo")
plt.ylabel("Periodo [s]")
plt.plot(periodos, "o")
[<matplotlib.lines.Line2D at 0x7fd0473084d0>]
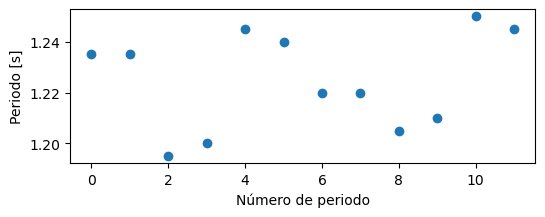
Para estos datos, es razonable calcular un periodo promedio.
Nota
Hay mejores formas de utilizar estos datos, pero probablemente exceda lo explicado cuando se realiza este experimento por primera vez.
np.mean(periodos)
np.float64(1.2249999999999999)
¿Qué error le asignaría a este promedio?
Analizando muchos archivos#
Como nombramos cada archivo con la condición experimental:
archivos[0] # el primer archivo
PosixPath('2cm.txt')
podemos extraerla de ahí:
extrar_longitud(archivos[0])
2.0
Esto nos facilita mucho al querer analizar muchas mediciones.
Por ejemplo, si resumimos los periodos en un promedio, podemos hacer una función que nos devuelva el periodo promedio y la longitud correspondiente.
def analisis(archivo):
t, v = cargar_archivo_de_motionDAQ(archivo)
tiempos = encontrar_tiempos_de_saltos(t, v)
periodos = calcular_periodos(tiempos)
periodo_promedio = np.mean(periodos)
longitud = extrar_longitud(archivo)
return longitud, periodo_promedio
analisis(archivos[0])
(2.0, np.float64(1.2249999999999999))
Y repetir esto para todos los archivos:
longitudes = []
periodos = []
for archivo in archivos:
L, T = analisis(archivo)
longitudes.append(L)
periodos.append(T)
longitudes = np.array(longitudes)
periodos = np.array(periodos)
longitudes
array([2., 1.])
periodos
array([1.225, 1.225])
Y graficar como varia el periodo con la longitud:
plt.figure(figsize=(6, 2))
plt.xlabel("Longitud [cm]")
plt.ylabel("Periodo [s]")
plt.plot(longitudes, periodos, "o--")
[<matplotlib.lines.Line2D at 0x7fd0a3448f50>]
También se puede hacer así:
resultados = np.array([analisis(archivo) for archivo in archivos])
resultados
array([[2. , 1.225],
[1. , 1.225]])
y después tomar cada columna por separado:
resultados[:, 0]
array([2., 1.])

